Академический Документы
Профессиональный Документы
Культура Документы
Exercice Corriger de Génie Génitique
Загружено:
Stephen Wright94%(17)94% нашли этот документ полезным (17 голосов)
4K просмотров11 страницАвторское право
© © All Rights Reserved
Доступные форматы
PDF, TXT или читайте онлайн в Scribd
Поделиться этим документом
Поделиться или встроить документ
Этот документ был вам полезен?
Это неприемлемый материал?
Пожаловаться на этот документАвторское право:
© All Rights Reserved
Доступные форматы
Скачайте в формате PDF, TXT или читайте онлайн в Scribd
94%(17)94% нашли этот документ полезным (17 голосов)
4K просмотров11 страницExercice Corriger de Génie Génitique
Загружено:
Stephen WrightАвторское право:
© All Rights Reserved
Доступные форматы
Скачайте в формате PDF, TXT или читайте онлайн в Scribd
Вы находитесь на странице: 1из 11
Exercice 1
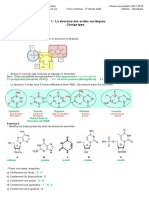
1) Identifier les bases prsentes dans les structures suivantes :
A = adnosine et guanine, B = uracile, C = cytosine, D = thymine
2) Parmi ces bases, lesquelles :
a) contiennent du ribose. B
b) contiennent du dsoxyribose. A, C
c) contiennent une purine. A
d) contiennent une pyrimidine B, C, D
e) contiennent de la guanine. A
f) sont des nuclosides. B
g) sont des nuclotides. A (dinuclotide), C
h) se trouvent dans lARN. B
i) se trouvent dans lADN. A, C, D
3) indiquer les extrmits 5 et 3 de la molcule A
5
3
et ceci sur la base de la reprsentation schmatique suivante de la structure de lADN :
ADN
ARN
p
o
l
y
a
n
i
o
n
s
Exercice 2
La squence dun ADN bicatnaire (double brin), correspondant un gne, est partiellement
reporte ci-dessous.
5 ATACGGGATCCGAGCTCTCGATCGTCTGCAGAAATTCC 3
1) Ecrire la squence et lorientation du second brin de ce fragment.
3 TATGCCCTAGGCTCGAGAGCTAGCAGACGTCTTTAAGG 5
Soit si lon respecte les conventions dcriture :
5 GGAATTTCTGCAGACGATCGAGAGCTCGGATCCCGTAT 3
2) Donner le brin complmentaire dARN
Luracile remplace la thymine
3 UAUGCCCUAGGCUCGAGAGCUAGCAGACGUCUUUAAGG 5
Soit : 5 GGAAUUUCUGCAGACGAUCGAGAGCUCGGAUCCCGUAU 3
3) Sur une reprsentation dtaille de lenchanement de deux nuclotides dun brin dADN du
site BamH I dont les bases seront reprsentes par les lettres correspondantes, indiquer ( laide
dune flche) quelle liaison est rompue sous laction de lenzyme BamH I.
Axe de symtrie
On rappelle que la spcificit des endonuclases de restriction est de :
! Reconnaitre des squences de bases spcifiques dans la double hlice dADN
- 4 15 paires de bases
- usuellement des palidromes
! Cliver les deux brins du duplex en des endroits trs spcifiques par hydrolyse de la liaison phosphodiester
pour obtention dun fragment de restriction
4) Soient les enzymes de restriction BamH I, Pst I, Xho I et Mbo I dont les sites reconnus sont :
BamH I : 5' G/ GATCC 3' ; Pst I : 5' CTGCA/ G 3' ; Xho I : 5' C/ TCGAG 3' ; Mbo I : 5' / GATC 3'.
Recopier la squence de lADN et encadrer les sites de restriction en indiquant la position des
coupures.
en surlign, les sites reconnus respectivement par les enzymes avec en noir, la coupure
correspondante
5 ATACGGGATCCGAGCTCTCGATCGTCTGCAGAAATTCC 3
3 TATGCCCTAGGCTCGAGAGCTAGCAGACGTCTTTAAGG 5
BamHI MboI Pst I
BamH I : 5' G/GATCC 3
3 CCTAG/G 5
Pst I : 5 CTGCA/G 3
3 G/ACGTC 5
Xho I : 5 C/TCGAG 3
3 GAGCT/C 5
Xho I ne reconnat pas de site sur ce fragment dADN donc naura pas daction (attention
lorientation du fragment dADN)
Mbo I : 5 /GATC 3'.
3 /CTAG 5
5) Pour chaque enzyme, crire les squences des extrmits des molcules dADN digres et
prciser le type dextrmits obtenu.
Pour BamH I (terminaisons bouts collants)
Fragment 1 : Fragment 2 :
5 ATACGG GATCCGAGCTCTCGATCGTCTGCAGAAATTCC 3
3 TATGCCCTAG GCTCGAGAGCTAGCAGACGTCTTTAAGG 5
Pour Pst I (terminaisons bouts collants)
Fragment 1 : Fragment 2 :
5 ATACGGGATCCGAGCTCTCGATCGTCTGCA GAAATTCC 3
3 TATGCCCTAGGCTCGAGAGCTAGCAG ACGTCTTTAAGG 5
Pour Mbo I (terminaisons bouts francs)
Fragment 1 : Fragment 2 :
5 ATACGGGATCCGAGCTCTC GATCGTCTGCAGAAATTCC 3
3 TATGCCCTAGGCTCGAGAG CTAGCAGACGTCTTTAAGG 5
6) On mlange ce brin dADN appari avec son brin complmentaire un autre fragment
dADN double brin. La solution est porte une temprature suprieure leurs Tm respectives,
puis refroidie. Que peut-on attendre?
Les brins dissocis (dnaturs) dADN de chaque espce vont se rapparier (se renaturer) avec
leur squence complmentaire sans mlange dADN des deux espces. Lappariement des bases
complmentaires est le plus stable nergtiquement.
8) Voici la squence dune amorce (ou primer) : 5 - TTTCTGCA- 3
O cette amorce se fixera-t-elle sur la squence dADN ? Quelle squence obtiendra-t-on aprs
longation par la DNA-polymrase ?
3 -ACGTCTTT- 5
5 ATACGGGATCCGAGCTCTCGATCGTCTGCAGAAATTCC 3
En vert, site reconnu par lamorce
En turquoise, lamorce
La DNA polymerase permet lajout dun dNTP (nuclotides triphosphates) lextrmit
hydroxyle en 3 de lADN :
3 TATGCCCTAGGCTCGAGAGCTAGCAGACGTCTTTAAGG 5
3 TATGCCCTAGGCTCGAGAGCTAGCAGACGTCTTT- 5
5 ATACGGGATCCGAGCTCTCGATCGTCTGCAGAAATTCC 3
En rose, squence obtenue aprs longation
Exercice 3
L'ADN dun cosmide recombinant* de 48,6 kpb a t hydrolys par l'enzyme de restriction Sal I.
Cet ADN est analys par lectrophorse sur gel dagarose (pistes 2 et 4). Un marqueur de taille
(l'ADN du phage lambda hydrolys par l'enzyme de restriction Hind III ) est dpos dans les puits
1 et 3. Ce marqueur de taille est un mlange quimolaire de fragments dADN de tailles connues.
La quantit dADN total dpos dans les puits 1 et 2 est le double de celle des puits 3 et 4. Aprs
coloration par le bromure dethidium**, limage suivante est obtenue.
* On rappelle quun cosmide est un vecteur artificiel constitu d'un plasmide auquel a t ajout le site COS du
bactriophage lambda. L'intrt des cosmides rside dans le fait qu'ils permettent le clonage de fragments d'ADN de
grande taille soit environ de 45kpb.
** Le bromure d'thidium (BET) une molcule aromatique plane qui s'intercale entre les paires de bases de la
double hlice et devient fluorescent. Il suffit ensuite d'clairer le gel avec une lumire ultra-violette (vers 310 nm)
pour voir la fluorescence orange du complexe ADN-BET dans les bandes.
1) laide des valeurs du tableau I, tracer la courbe talon :
Log taille(pb) = f(distance de migration).
Type de courbe obtenir :
2
2.5
3
3.5
4
4.5
0 5 10 15 20
2) partir de cette courbe, dduire la taille des fragments issus de la digestion par l'enzyme de
restriction Sal I de l'ADN du cosmide recombinant (pistes 2 et 4).
Migration
(cm)
Taille
(pb)
2.8 14692
3.2 11434
3.6 9165
4.1 7179
6.5 3021
SOMME 45490
3) La somme des tailles des fragments obtenus vous semble-t-elle en accord avec la taille
attendue de 48,6 kpb ?
La somme est plus faible. Cela peut tre d notamment aux incertitudes qui peuvent tre
importantes en particulier pour les fragments de grande taille (voir allure courbe) ou deux
bandes trs proches qui nauraient pas t diffrencies la lecture.
4) Le bromure dethidium s'intercale entre les bases de l'ADN de manire uniforme sur une
molcule d'ADN linaire. tudiez attentivement la photo du profil de restriction, que remarquez-
vous ? Quen dduisez-vous ?
La fluorescence des bandes 1 et 2 est plus intense. Or la quantit dADN total dpos dans les
puits 1 et 2 est le double de celle des puits 3 et 4. La lecture du gel permet donc, au travers de
lintensit de la fluorescence mise, dobtenir une information semi-quantitative et de comparer
de manire relative les quantits dADN au niveau de chaque bande.
Exercice 4 : Clonage et analyse de l'ADN recombinant
On souhaite tudier la fonctionnalit dun gne M dune bactrie. Pour cela, on essaie de cloner
au site Eco RI du vecteur plasmidique pBR330 (voir schma) un fragment Eco RI-Eco RI d'ADN
gnomique de la bactrie dintrt :
a- Proposez un protocole de clonage et indiquez comment vous slectionnez les clones
recombinants.
! Prparation de l'insert: Extraction d'ADN gnomique de la bactrie. Digestion
partielle de cet ADN par l'enzyme de restriction Eco RI, de manire gnrer des
fragments de 5 10 kb. Electrophorse prparative pour purifier sur gel les fragments de
5 10 kb.
! Prparation du vecteur: Digestion complte de pBR330 par Eco RI.
Dphosphorylation des extrmits.
! Clonage: ligature des inserts et des vecteurs. Transformation de cellules d'E. coli.
Recommencer les tapes prcdentes jusqu' avoir 10 e+6 colonies transformes
indpendantes.
! Slection: slection des colonies transformes sur milieu contenant de la streptomycine.
Pour estimer le taux de colonies qui ont reu un plasmide contenant de l'ADN gnomique
de la bactrie tudie, repiquage d'environ 500 colonies sur milieu contenant de
l'ampicilline: seule les colonies qui ont reu un plasmide ne contenant pas d'ADN
gnomique de la bactrie tudie poussent.
! Slection des clones contenant le gne dintrt: transfert des 10 e+6
colonies sur filtre et hybridation molculaire avec une sonde approprie (technique dite de
"l'hybridation sur colonies").
b- Un des plasmides recombinants contenant le gne M (appel pBM1) est digr par les
enzymes de restriction Bam HI et Eco RI. Aprs migration et sparation des fragments d'ADN sur
gel d'agarose puis coloration au bromure d'thidium, on obtient les profils de restriction suivants:
Donnez la carte de restriction du plasmide recombinant pBM1.
Exercice 5 : Squenage
Vous disposez dun brin dADN squencer (matrice), dune amorce, dADN polymrase
tronque*, des quatre 2-dsoxyribonuclotides (dXTP) et dun jeu des diffrents 2,3-
didsoxyribo-nuclotides (ddXTP). Lamorce est radiomarque par du phosphore radioactif
32
P.
LADN matriciel squencer ACGTAATCGC---- comporte, son extrmit 3, une squence
supplmentaire (reprsente ici par un segment de droite en pointill) sur laquelle lamorce va
shybrider, crant ainsi le site dinitiation de lADN polymrase.
*lenzyme est constitue de plusieurs domaines dont lun est responsable de la destruction " programme " de
lamorce ; aprs limination de ce domaine, la fraction restante, dsigne " fragment de Klenow ", conserve
lactivit ADN polymrase.
1 Rsumez brivement le principe de la mthode en indiquant le rle du
didsoxyribonuclotide.
Loriginalit du ddXTP (fluorescent ou radioactif) dans la mthode utilise (Mthode de Sanger)
est quil stoppe la progression de la synthse (absence du groupement OH)
.
Dsoxyribonuclotide
triphosphate
(dXTP)
Didsoxyribonuclotide
triphosphate
(ddXTP)
Rappel de la mthode :
1 la rplication (ou duplication) de lADN ncessite la prsence dun brin modle (matrice),
dune amorce complmentaire dun fragment dADN qui jouxte la rgion squencer, des 4
nuclotides dXTP, dADN polymrase.
2 la synthse du brin complmentaire se faisant dans la direction 5P vers 3OH, le brin
matriciel a lorientation inverse ; lamorce se lie du ct 3 de lADN matriciel ; lADN
polymrase parcourt ce brin dans sa direction 3-5 pour allonger lamorce dans la direction
correcte 5-3.
3 lorsque lADN polymrase identifie A sur le brin modle, elle place T sur le brin en cours de
synthse (de mme pour C et G).
Quand des ddXTP sont utiliss :
Lenzyme place soit 1 dXTP et llongation de lamorce se poursuit, soit 1 ddXTP et la synthse
sarrte. On obtient au final une srie doligonuclotides de taille variable, tous termins par
ddX (X = A, G, C, T selon le milieu). Le rsultat de llectrophorse des 4 milieux donne
directement la squence de lADN complmentaire la squence recherche.
2 Complter le tableau en indiquant la composition des diffrents milieux ractionnels et, pour
chaque milieu, le type et la taille des fragments nosynthtiss.
3 Sur le gel ci-dessous, reprsenter la taille des fragments nosynthtiss dans chaque milieu
ractionnel (utiliser lchelle de taille reprsente gauche du schma)
4 Reporter, droite, la squence du brin synthtis puis la squence recherche, en indiquant
le sens de lecture des squences tablies.
Exercice 6 : Squenage
Un petit fragment dADN a t squenc selon la mthode dinterruption des chanes (mthode
de Sanger). Une fois la raction de squence termine, la taille des fragments obtenus est
dtermine par une chromatographie. Le squenceur automatique pourvu dune source laser ou
infra-rouge qui excite les fluorochromes ports par les ddNTP, dtecte la fluorescence sortant des
colonnes de chromatographie, reprant ainsi les fragments d'ADN et leur taille prcise. Le
rsultat est prsent sous forme de courbes prsentant la fluorescence dtecte, et l'interprtation
qui en faite en terme de nuclotidesDonner la squence de lADN (la flche sur le dessin indique
le sens de migration)
Des squences de 300 nuclotides sont squences de manire correcte sur gel ( la main ).
Les squenceurs permettent de lire plusieurs centaines de nuclotides avec une trs bonne qualit
et ceci jusqu' 1000
Вам также может понравиться
- Mistral GagnantДокумент5 страницMistral GagnantLucas Joan Margenats Lynch50% (2)
- Exercices de Génie GénétiqueДокумент6 страницExercices de Génie GénétiqueNadineHana91% (32)
- Correction TD 305 PDFДокумент28 страницCorrection TD 305 PDFJalelo AbdelОценок пока нет
- Exercice de GénétiqueДокумент69 страницExercice de GénétiqueOumarou KontaОценок пока нет
- TD-BM 2012Документ50 страницTD-BM 2012KhalilUchiha95% (21)
- QCMДокумент7 страницQCMSarah SaRiTa100% (1)
- Cahier D'exercice4Документ28 страницCahier D'exercice4GHERMI .M100% (6)
- Biologie Molecule TD CorrigéДокумент3 страницыBiologie Molecule TD CorrigéSchola Janvier94% (31)
- TD Génétique L2Документ33 страницыTD Génétique L2Maroua Sarah Bellel79% (14)
- 3 La RéplicationДокумент17 страниц3 La Réplicationbahidjakheira belouadi100% (1)
- Td1 Corrige TypeДокумент3 страницыTd1 Corrige TypeLina BiologistaОценок пока нет
- ReplicationДокумент21 страницаReplicationMohamed Yassine MtarОценок пока нет
- Contrôle Techniques 2019Документ7 страницContrôle Techniques 2019Sam Épse Rbi100% (1)
- Génomique CorrigéДокумент38 страницGénomique CorrigéHicham mesbahОценок пока нет
- E9782729831967 PDFДокумент8 страницE9782729831967 PDFأبو رضا100% (2)
- Corrigé Sérien°3 S2-2012Документ11 страницCorrigé Sérien°3 S2-2012Mind Opnd100% (1)
- Examen 28 Mai 2010-CorrigéДокумент6 страницExamen 28 Mai 2010-Corrigéikram bt0% (1)
- Exercices Enzym RestrictionДокумент17 страницExercices Enzym Restrictionkhmaj979295% (55)
- 1er Cours Introduction Au Monde MicrobienДокумент8 страниц1er Cours Introduction Au Monde MicrobienMoufida Trénitarya100% (1)
- 3 Serie Td3 La Synthese Des Proteines 2016 2017Документ2 страницы3 Serie Td3 La Synthese Des Proteines 2016 2017Houleye Kane100% (4)
- Corrige-Type td1 Genie Genetique 3eme Annee LMD BiochimieДокумент2 страницыCorrige-Type td1 Genie Genetique 3eme Annee LMD Biochimieamina44100% (3)
- Génie Genet. Exercices - 22-23Документ36 страницGénie Genet. Exercices - 22-23Hanae Benhaida100% (1)
- Genetique1an16-Biologie Moleculaire Genie GenetiqueДокумент34 страницыGenetique1an16-Biologie Moleculaire Genie Genetiqueikram bt100% (2)
- TD GENIE GENETIQUE Série 3Документ3 страницыTD GENIE GENETIQUE Série 3meriama deb100% (5)
- Mycoplasmes DR GonsuДокумент44 страницыMycoplasmes DR GonsuIdrissou FmsbОценок пока нет
- Southern Blot-Hybridation-SondeДокумент51 страницаSouthern Blot-Hybridation-Sondeyouness.khalfaoui100% (4)
- Biologie Moléculaire Examens 01 PDFДокумент10 страницBiologie Moléculaire Examens 01 PDFbiotech micro0% (1)
- LV203 Sujet Et Corrige 1er CC 2006Документ4 страницыLV203 Sujet Et Corrige 1er CC 2006Za Hra100% (1)
- TD #5 PCR - CorrigéДокумент4 страницыTD #5 PCR - CorrigéNihal Krika100% (6)
- Ex Carte de RestrictionДокумент19 страницEx Carte de RestrictionIMAD100% (2)
- S3F1. P1. Exercices Dirigés + CorrigéДокумент7 страницS3F1. P1. Exercices Dirigés + CorrigéAnonymous MKSfyYyODP100% (5)
- TD Corrigé de Biologie MoléculaireДокумент3 страницыTD Corrigé de Biologie MoléculaireGeronimo0% (1)
- TD Génie GénétiqueДокумент4 страницыTD Génie GénétiqueMeryem ELkhalloufy100% (1)
- ExercicesДокумент6 страницExercicesNina Bouredji100% (4)
- TD 1 Corrigé GénétiqueДокумент5 страницTD 1 Corrigé GénétiqueDoudou AmineОценок пока нет
- Biomol Exam 2Документ5 страницBiomol Exam 2Amina GaamoussiОценок пока нет
- BG3 ExosДокумент22 страницыBG3 Exospersonne12Оценок пока нет
- Series TD GenetiqueДокумент36 страницSeries TD GenetiqueNouhoum TraoréОценок пока нет
- Biologie MoléculaireДокумент10 страницBiologie Moléculairemalak wxОценок пока нет
- Corrigé TD 3Документ3 страницыCorrigé TD 3kim namjoon100% (2)
- Poly TD BIO MOL 2Документ11 страницPoly TD BIO MOL 2arminОценок пока нет
- 2) Exercices Structure Et Organisation de l'ADNДокумент17 страниц2) Exercices Structure Et Organisation de l'ADNOne Love LifeОценок пока нет
- Controle 1 Et 2 Bio MoléculaireДокумент6 страницControle 1 Et 2 Bio Moléculaire[AE]100% (4)
- TD 6 Avec CorrigéДокумент2 страницыTD 6 Avec CorrigéAbdellatif Abdrahim100% (1)
- TD GENIE GENETIQUE Série 4Документ2 страницыTD GENIE GENETIQUE Série 4meriama deb100% (1)
- TD RecapitulatifДокумент4 страницыTD RecapitulatifZa Hra100% (2)
- Vecteurs de ClonageДокумент37 страницVecteurs de Clonagelobna75% (4)
- Acides Nucleiques PDFДокумент90 страницAcides Nucleiques PDFZineb DahmaniОценок пока нет
- Corrigé Type Du TD1 Génétique (La Structure Des Acides Nucléiques)Документ3 страницыCorrigé Type Du TD1 Génétique (La Structure Des Acides Nucléiques)Doudou Amine50% (2)
- Emd 08Документ8 страницEmd 08lmd2009Оценок пока нет
- L3 Biochimie 19-20 Messaoudi D L3 Bioch TD 3 Corrigé Biologie MoléculaireДокумент2 страницыL3 Biochimie 19-20 Messaoudi D L3 Bioch TD 3 Corrigé Biologie MoléculaireYassine RiahiОценок пока нет
- Fiche de TD1 2016 2017-1Документ3 страницыFiche de TD1 2016 2017-1Berthe Aristide Ngue Nkongo100% (3)
- Transferts Génétiques Chez Les BactériesДокумент5 страницTransferts Génétiques Chez Les BactériesZa Hra50% (2)
- Cours TD Gen ProДокумент51 страницаCours TD Gen Prolobna67% (3)
- Series de TD Module BIBДокумент22 страницыSeries de TD Module BIBCamel CamelinОценок пока нет
- TD 2 D'enzymologieДокумент3 страницыTD 2 D'enzymologieabdel krim100% (2)
- Corrige-Type td2 Genie Genetique 3eme Annee LMD BiochimieДокумент4 страницыCorrige-Type td2 Genie Genetique 3eme Annee LMD Biochimieamina44Оценок пока нет
- T.D 1 2° LMD-GénétiqueДокумент2 страницыT.D 1 2° LMD-GénétiqueMohamed Badreddine Mokhtari100% (1)
- TD2 Biologie MoléculaireДокумент7 страницTD2 Biologie Moléculairejebranesanaa92Оценок пока нет
- TD de BM Et Génétique No1Документ8 страницTD de BM Et Génétique No1habessongОценок пока нет
- Travaux Dirigés de Génie - GénétiqueДокумент13 страницTravaux Dirigés de Génie - Génétiquedombuyvan123Оценок пока нет
- $TD #1 D O.B PDFДокумент2 страницы$TD #1 D O.B PDFNacera CeraОценок пока нет
- Série 04Документ3 страницыSérie 04zakibelarbi018Оценок пока нет
- Clonage PDFДокумент37 страницClonage PDFJalelo AbdelОценок пока нет
- TD Bio Cell 1ère MedДокумент65 страницTD Bio Cell 1ère Medouma imaОценок пока нет
- Biologie Moléculair0 PDFДокумент44 страницыBiologie Moléculair0 PDFDoudou SamiОценок пока нет
- These Benabdelkader Tarek 2012Документ281 страницаThese Benabdelkader Tarek 2012Kima MadОценок пока нет
- Expose Fer3Документ13 страницExpose Fer3vague2000Оценок пока нет
- Reponse S Biolog I e 100712Документ7 страницReponse S Biolog I e 100712steve_620Оценок пока нет
- 08 HyaloplasmeДокумент2 страницы08 HyaloplasmeILHEM RAMDAОценок пока нет
- Ble Tendre Ou Ble DurДокумент3 страницыBle Tendre Ou Ble DuraliciaОценок пока нет
- Classification Raisonnée Des Bétalactamases Des Gram NégatifsДокумент38 страницClassification Raisonnée Des Bétalactamases Des Gram NégatifsFlora08Оценок пока нет
- Sujet CorrigeДокумент7 страницSujet Corrigeh3r3t1k100% (1)
- UntitledДокумент398 страницUntitledMEDОценок пока нет
- Seve Michel p02Документ16 страницSeve Michel p02Manel YahiaouiОценок пока нет
- ECOPEgenpop Exo 3Документ7 страницECOPEgenpop Exo 3منصور الأمجدОценок пока нет
- ABT-hopital-2009 - Durpi PDFДокумент58 страницABT-hopital-2009 - Durpi PDFHuplala HypocrateОценок пока нет
- Canevas Master Harmonisé Biochimie AppliquéeДокумент56 страницCanevas Master Harmonisé Biochimie AppliquéeMouhi MouhaОценок пока нет
- Le MicrobeДокумент27 страницLe MicrobemarwenОценок пока нет
- Physio1an-Interaction Ligand Recepteur2016Документ18 страницPhysio1an-Interaction Ligand Recepteur2016AmirMohamedYozmaneОценок пока нет
- Intro GenomiqueДокумент51 страницаIntro Genomiquefatima zohra mahammiОценок пока нет
- SVT Obligatoire S PDFДокумент5 страницSVT Obligatoire S PDFLetudiant.frОценок пока нет